Orbitrap多种碎裂方式深度解析脂质精细结构
发布时间:2025-07-23 09:47 | 点击次数:339
脂质是生物体重要的结构基石和功能分子。以甘油磷脂、胆固醇为核心的脂质分子组成细胞膜双分子层结构,为生命活动提供稳定的物理屏障和微环境;而甘油三酯直接参与能量动员和新陈代谢,神经酰胺、类花生酸和磷脂酰肌醇是重要的信号转导介质。因此,全面表征脂质结构有助于揭示脂质与其他生物分子复杂的功能网络。
脂质的精细结构表征通常包含如下多个层级的结构信息[1,2]:1) 确定脂质种类(主要通过识别脂质的头基);2)确定脂质亚类(通过识别脂肪酸连接方式确认属于醚脂、缩醛磷脂或酰基脂等);3)脂质分子的总体脂肪酸水平;4)脂质分子各酰基脂肪酸水平;5)不饱和脂肪链碳碳双键(C=C)位置和立体构型、其他修饰(如甲基、环丙基、羟基等)位置;6)脂肪酸链sn位置(图1)。其中,高分辨质谱和串联质谱已经实现前四个水平的脂质结构鉴定,但对于表征脂质C=C和sn位置仍存在巨大挑战。
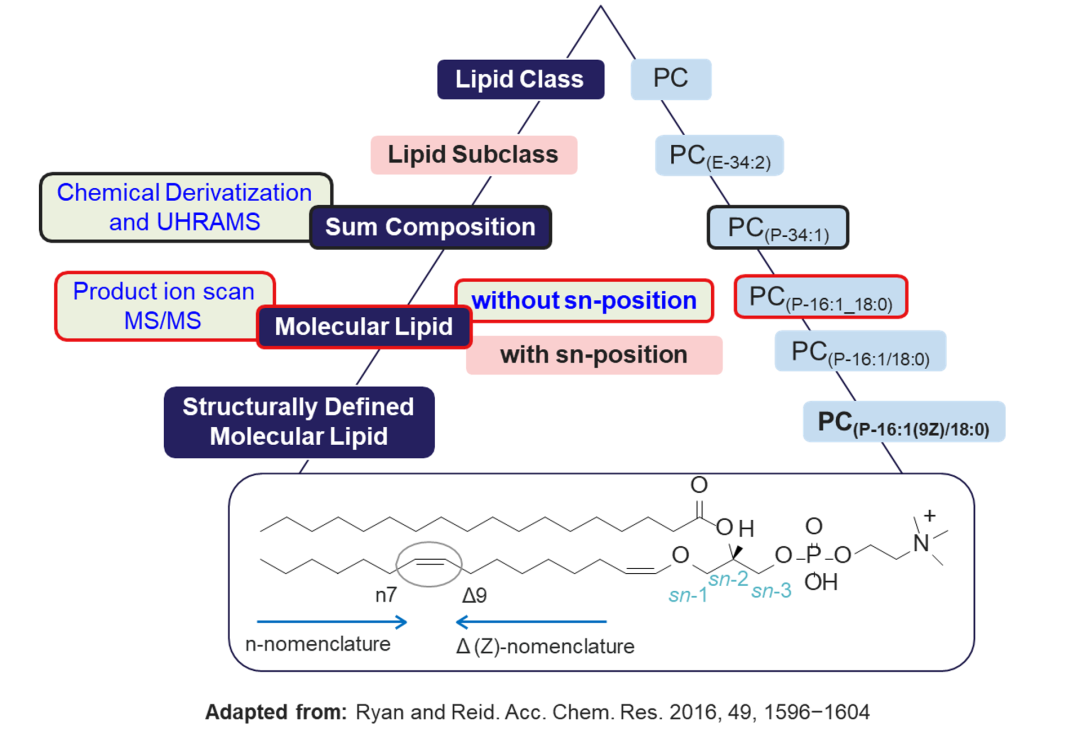
图1. 脂质分子的鉴定水平(点击查看大图)
1
Thermo Scientific™ Orbitrap™ Tribrid™ 三合一质谱全面解析脂质结构
以磷脂酰胆碱(PC)为例,传统LC-MS分析中PC在正离子模式下只能得到头基离子(m/z 184.07)的碎片(图2上),并不能得到脂肪酸链的组成信息,因此通常需要结合负离子模式确定酰基组成。在Orbitrap™ Tribrid™ 三合一系列质谱中, 除了应用常规beam-type HCD碎裂得到头基信息外,还可以利用阱内CID(ion-trap CID)直接在一针正模式分析中获得完整脂肪酸组成信息,极大提高脂质组学的分析通量(图2下)。
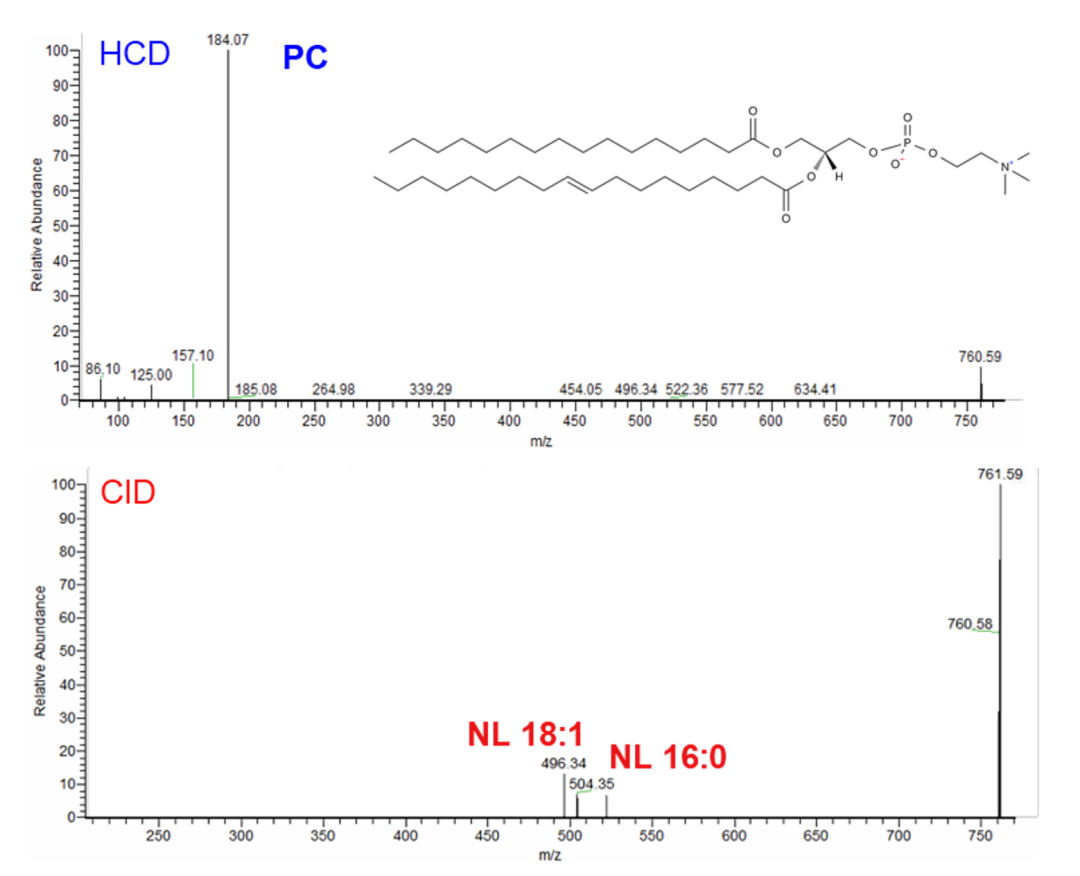
图2.PC(16:0/18:1)的HCD(上)和CID(下)正模式MS/MS谱图。在HCD模式下,只能观测到头基离子信号;而在CID谱图中,可以观测到FA(16:0)和FA(18:1)中性丢失离子,从而获得PC(16:0/18:1)分子组成。
(点击查看大图)
再如甘油三酯(TG)的结构解析中,二级质谱只能在中性丢失水平上推测TG三条脂肪酸链可能的组成信息, 但针对共流出的异构体物种缺乏分辨能力。例如虽然TG(48:1) MS2谱图可以通过FA(18:1), FA(16:0) 和FA (14:0) 三条酰基链中性丢失推断该TG为TG(14:0/16:0/18:1)。但利用Orbitrap三合一质谱的多级质谱分析功能,可以在MS3谱图层面轻松分辨出其中共流出的TG(16:1/16:0/16:0) 分子种,极大提高TG分子的鉴定深度(图3)。
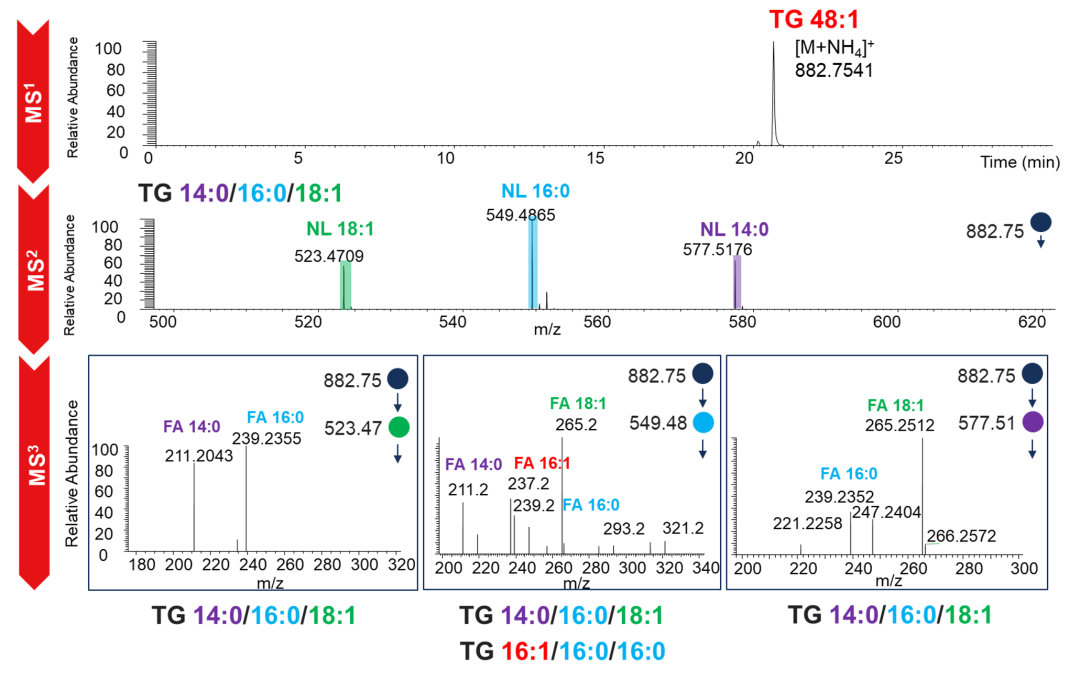
图3.TG(48:1)分子的结构表征。MS2需要通过三条脂肪链中性丢失离子的排列组合推断其分子组成,但对于共流出的异构体无分辨能力。而通过MS3分析可以进一步得到脂肪酸链诊断离子,特别是对共流出TG的鉴定具有重要作用(点击查看大图)
基于此,针对复杂脂质组学分析,我们在Orbitrap™ IQ-X™ Tribrid™ 三合一质谱中搭建了如图4所示的质谱分析模板用于非靶向脂质组学分析。首先对传统HCD-MS2谱图快速扫描,针对PC头基离子触发CID-MS2补充其脂肪链组成信息;针对TG的特定中性丢失触发HCD-MS3提供更精准的结构注释。
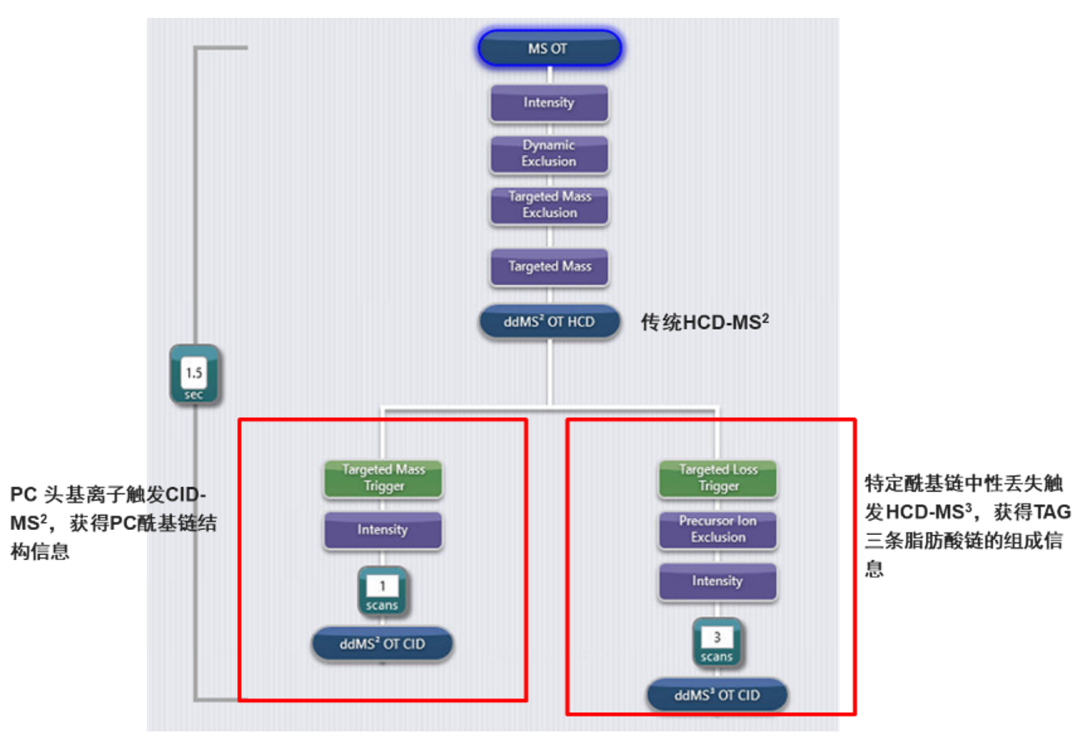
图4.完整脂质组学分析质谱模板
(点击查看大图)
2
UVPD精确表征甘油磷脂C=C位置
脂质的双键结构显著影响生物膜通透性和内环境动态平衡,大量研究证实表征脂质C=C位置有助于了解细菌耐药和肿瘤发生发展。脂质的C=C在200nm的波长范围内存在一定的紫外吸收,因此193nm和213nm的紫外光解离(UVPD)技术在脂质结构深度表征方面起到了重要作用。Thermo Scientific™ Orbitrap™ Tribrid™ 三合一系列质谱(如IQ-X,Ascend等型号)可附加UVPD模块用于脂质C=C位置的深度表征(图5)。该模块将213nm紫外光照射进入双压线性离子阱,被离子阱捕获的脂质C=C双键在吸收光子后发生π-π*跃迁进入激发态,诱导邻位C-C键断裂,形成一对相差24Da的诊断离子,从而指认C=C位置[2](图5)。
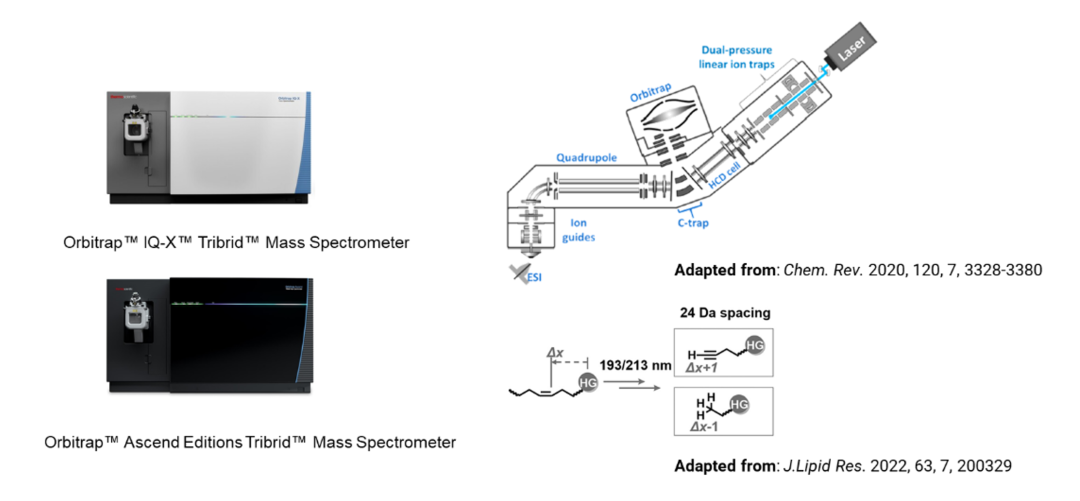
图5. UVPD可作为附加模块配置在Orbitrap™ IQ-X™等三合一系列质谱,UVPD工作原理以及分析C=C诊断离子
(点击查看大图)
以PC(16:0/18:1n-9)为例,在UVPD激发下C=C邻位C-C断裂,形成x+1炔离子和x-1烷基离子,这两个诊断离子分子量相差24Da,可用于C=C双键位置(x)的确认[4]。相比于其他碎裂方式,UVPD对C=C的碎裂更具有选择性,诊断离子附近无其他干扰离子,即使需要放大谱图查找,也极易识别(图6)。在真实的鼠脑组织中,UVPD成功鉴定出PC(18:1/18:1)的三种C=C异构体,包括PC(18:1 n-9/18:1 n-9)、PC(18:1 n-7/18:1 n-7)、PC(18:0/18:2n-6,9)等(图7)。
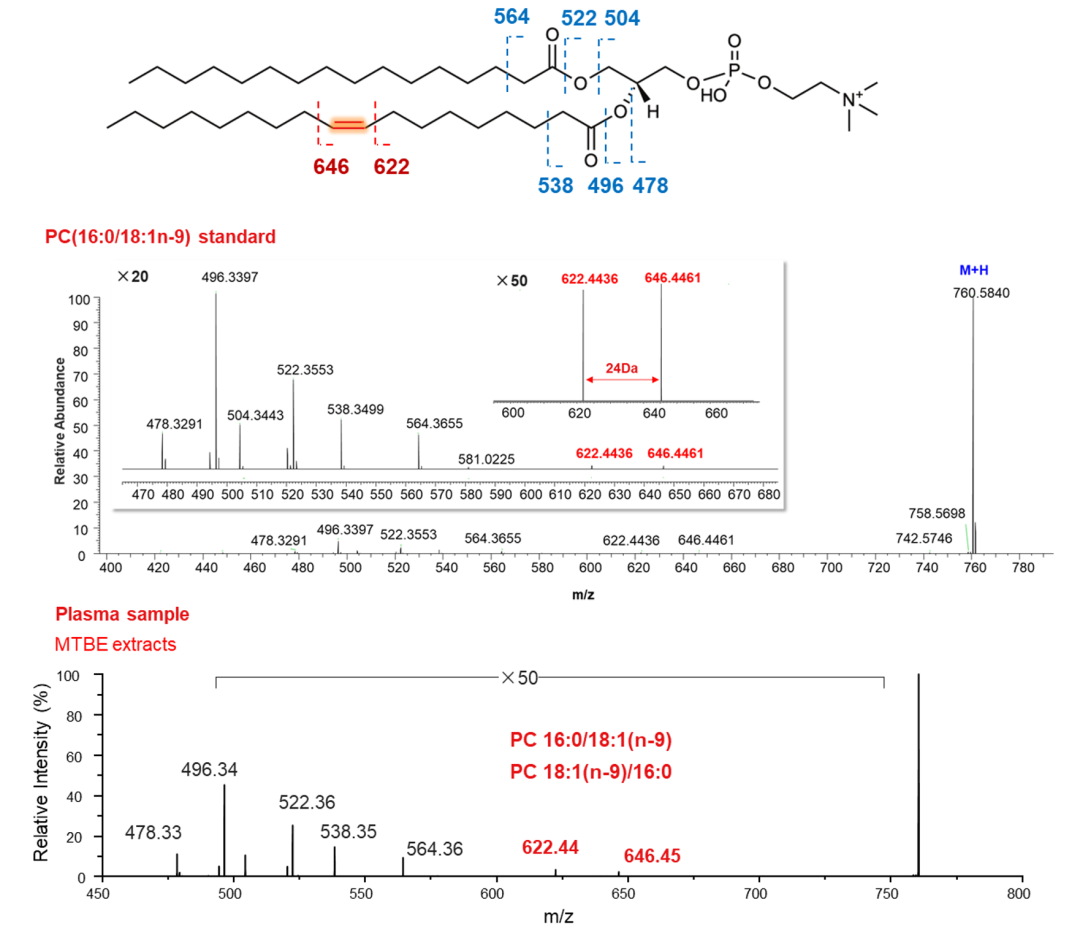
图6. PC(16:0/18:1n-9)标准品和血浆样本UVPD-MS2谱图。UVPD激发可以诱导C=C邻位断裂,形成质量数相差24Da的双键诊断离子(点击查看大图)
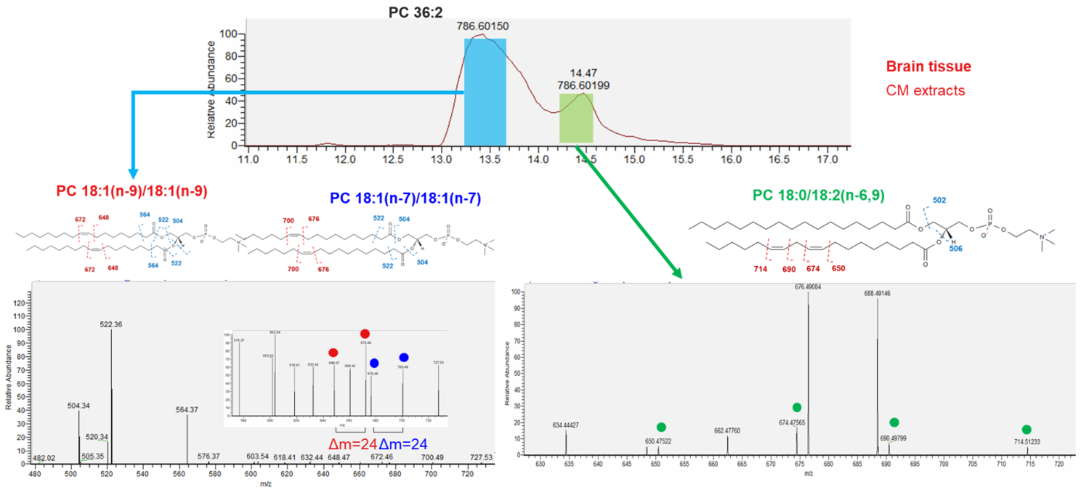
图7. 真实小鼠脑组织样本中PC(18:1/18:1)双键异构体鉴定和表征(点击查看大图)
滑动查看更多
3
UVPD 用于脂质sn位置的深度表征
磷脂的两条脂肪链的sn位置反映了脂质从头合成过程,但LC-MS通常难以分离sn异构体。磷脂酶A(PLA1和PLA2)选择性水解法虽然可以成功鉴定sn异构体,但检测通量较低。研究发现,磷脂酰胆碱的碱金属加合离子在HCD/CID解离后会丢掉头基离子,通过重排反应形成的二yang戊环结构,UVPD会特异性切断sn-2位置脂肪酸链,得到仅含sn-1脂肪酸链的诊断离子,因此可以用于磷脂和TG的sn-异构体鉴定[5]。UVPD-MS3无需放大谱图即可识别特征sn异构离子,可用于常规非靶向脂质组学分析工作流。
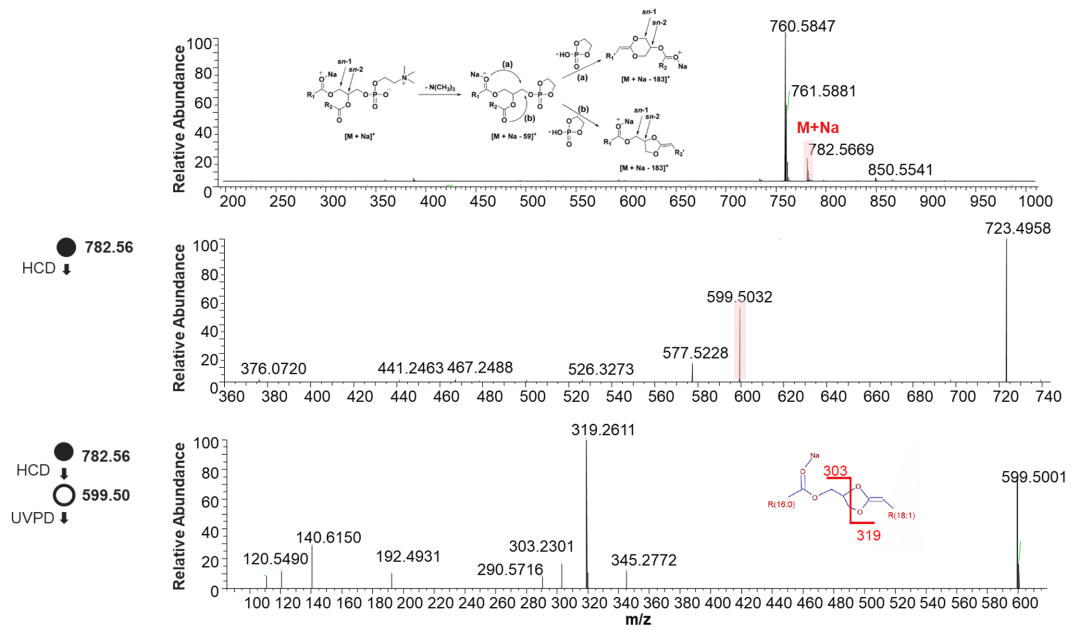
图8. PC(16:0/18:1)sn结构鉴定,选择碱金属加合离子[M+Na]+产生的HCD/CID-MS2谱图中[M+Na-183]+碎片离子,进一步触发UVPD-MS3,可以得到选择性sn-1脂肪链结构信息,从而推测PC的sn位置
(点击查看大图)
4
相关应用文献
(1)
UVPD全面表征甘油磷脂结构
甘油磷脂(GPLs)是细胞膜主要成分之一,显著影响细胞膜流动性和通透性,细菌通过调控甘油磷脂改变细胞膜表面电荷从而影响抗生素耐药。其中,磷脂的环丙基修饰与结核分枝杆菌耐药相关,但传统的CID/HCD技术无法表征环丙基结构的位置。考虑到环bing烷基在200nm波长附近也存在紫外吸收,德克萨斯大学奥斯汀分校的研究小组利用UVPD技术轻松诱导环丙基邻位C-C键断裂产生间隔14Da的诊断离子对,从而实现环丙基磷脂的精确定位,并在大肠杆菌和结核分枝杆菌中鉴定出一系列未曾报道的甘油磷脂环丙基修饰结构(图9上)。随后,该小组应用HCD/UVPD对细菌GPLs进行了综合、全面的结构解析,阐明了细菌脂质中不饱和键(碳碳双键、环丙基)、羟基和烷基等修饰与细菌耐药性的关系[5,6](图9下)。
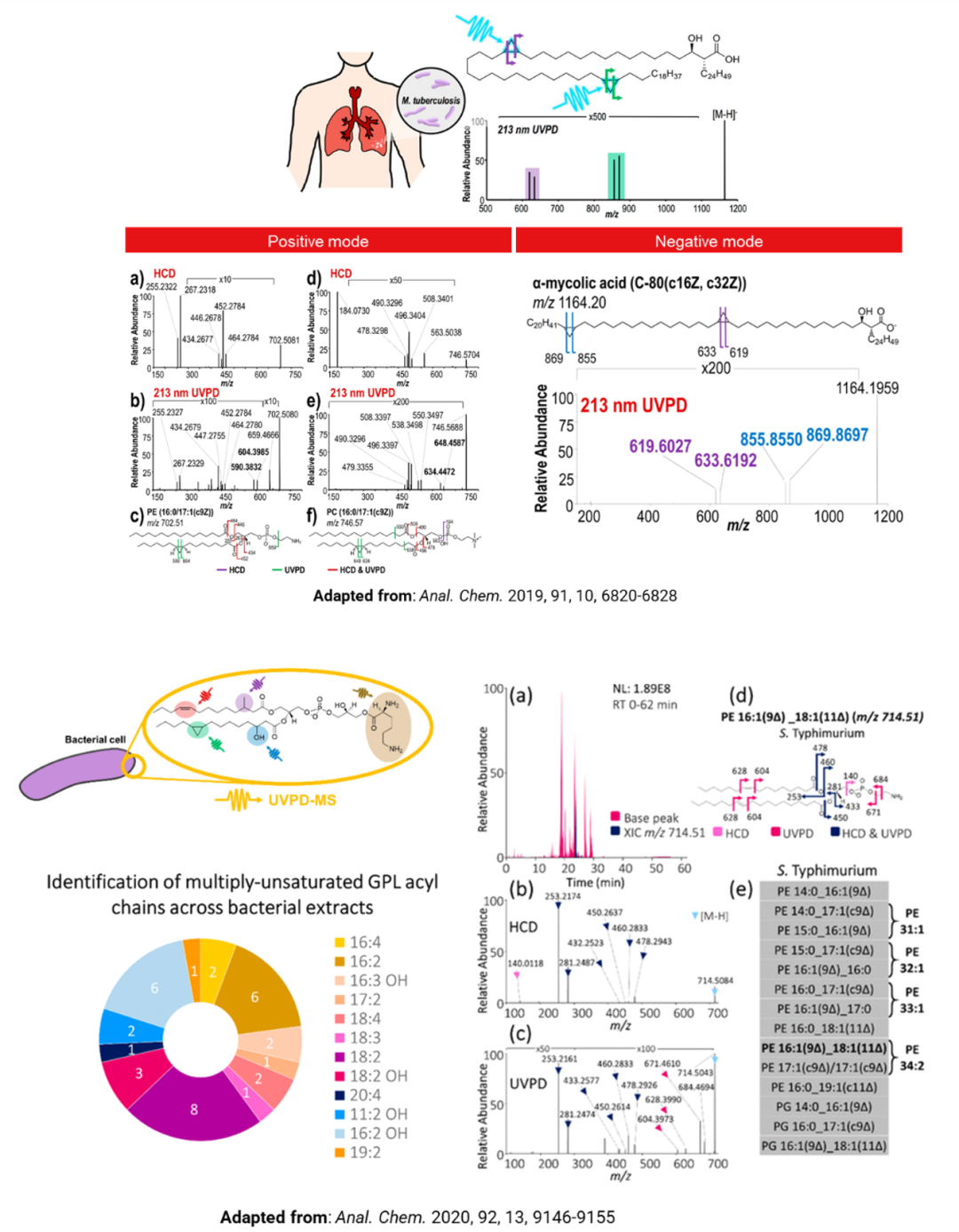
图9. 全面表征甘油磷脂结构,包括C=C位置、环丙基位置、羟基位置等,揭示甘油磷脂不饱和键种类和分布于细菌耐药机制的关系(点击查看大图)
(2)
其他重要脂质分子精细结构解析
除了在正离子模式下表征GPLs外,UVPD也成功应用于表征包括羟基脂肪酸酯[7]、鞘脂[8]、固醇[9]、糖脂[10,11]等脂质结构。例如,心磷脂含有四条酰基链,是一种复杂程度更高的脂质,采用混合碰撞(CID×UVPD)方法可以精确定位心磷脂四个酰基链碳碳双键和环丙基不饱和键的位置[12],而通过化学衍生化结合HCD/UVPD可以进一步解析心磷脂的sn-异构体[13],实现未知心磷脂结构的从头注释(图10)。除此之外,UVPD也被证实可以在负离子模式下应用于游离脂肪酸和磷脂酰甘油(PG)等结构表征[14]。
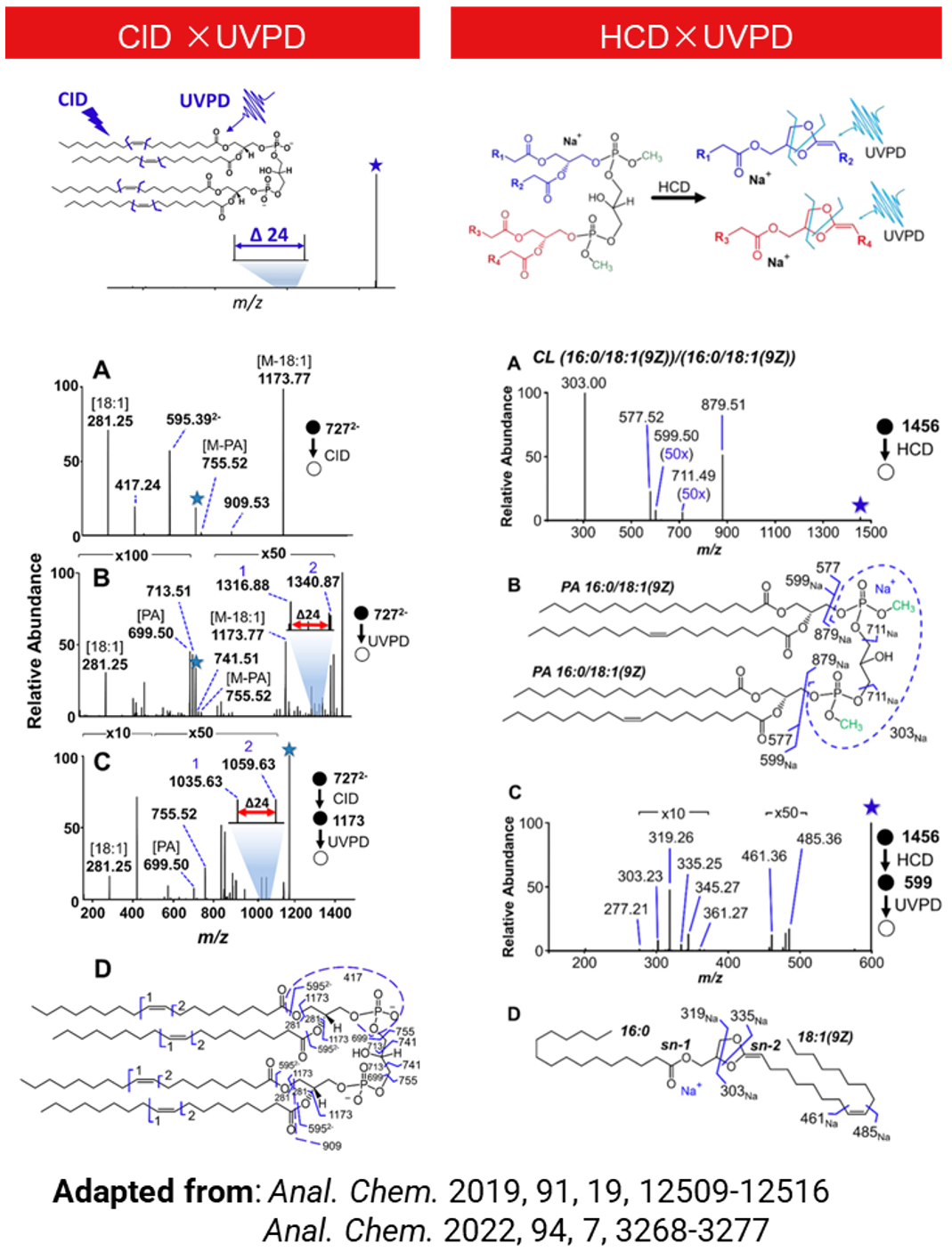
图10. CID/HCD与UVPD混合碎裂用于心磷脂C=C双键和sn位置的确认(点击查看大图)
(3)
DESI-UVPD用于不饱和磷脂的空间成像
解吸电喷雾电离(DESI)常用于脂质分子的空间成像,但传统MS1成像对同分异构体的识别能力显然不足。研究者利用UVPD活化脂质产生的C=C诊断离子强度(Im/z 660+684)/(Im/z 688+712) 反映PC 16:0_18:1(n-9),和PC 16:0_18:1(n-7)]在脑组织切片中的分布,实现癌组织和癌旁组织区域的病理勾画[15]。
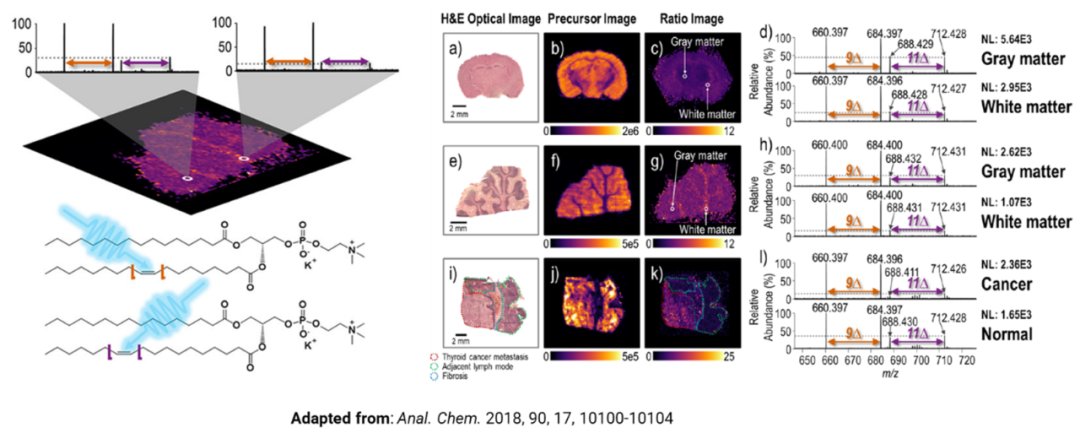
图11. DESI-UVPD-MS2用于脂质C=C异构体成像
(点击查看大图)
总 结
在生命科学研究中,脂质分子以其结构复杂性和动态多样性承载细胞膜和物理屏障构建、信号传递和能量代谢的核心使命。Thermo Scientific™ Orbitrap™ Tribrid™ 三合一系列质谱重塑脂质结构鉴定的通量和深度,其通过多级质谱解析和多重碎裂技术(HCD,CID,UVPD)突破脂质分析的极限,实现在碳碳双键、sn位置及酰基修饰模式等方面的深度洞察,从而终结脂质分子结构的模糊解析,更好地助力全球科学家在代谢脂质组学领域的科研突破。
参考文献:
1. Ryan E, Reid GE. Chemical Derivatization and Ultrahigh Resolution and Accurate Mass Spectrometry Strategies for "Shotgun" Lipidome Analysis. Acc Chem Res. 2016;49(9):1596-1604.
2. Zhang W, Jian R, Zhao J, Liu Y, Xia Y. Deep-lipidotyping by mass spectrometry: recent technical advances and applications. J Lipid Res. 2022;63(7):100219.
3. Macias LA, Garza KY, Feider CL, Eberlin LS, Brodbelt JS. Relative Quantitation of Unsaturated Phosphatidylcholines Using 193 nm Ultraviolet Photodissociation Parallel Reaction Monitoring Mass Spectrometry. J Am Chem Soc. 2021;143(36):14622-14634.
4. Williams PE, Klein DR, Greer SM, Brodbelt JS. Pinpointing Double Bond and sn-Positions in Glycerophospholipids via Hybrid 193 nm Ultraviolet Photodissociation (UVPD) Mass Spectrometry. J Am Chem Soc. 2017;139(44):15681-15690.
5. Blevins M S, Klein D R, Brodbelt J S, Localization of cyclopropane modifications in bacterial lipids via 213 nm ultraviolet photodissociation mass spectrometry. Anal Chem, 2019, 91:6820-6828.
6. Blevins M S, James V K, Herrera C M, et al., Unsaturation elements and other modifications of phospholipids in bacteria: New insight from ultraviolet photodissociation mass spectrometry Anal Chem, 2020, 92:9146-9155.
7. Buenger E W, Reid G E. Shedding light on isomeric FAHFA lipid structures using 213 nm ultraviolet photodissociation mass spectrometry. Eur J Mass Spectrom, 2020,26(5):311-323.
8. Ryan E, Nguyen CQN, Shiea C, Reid GE. Detailed Structural Characterization of Sphingolipids via 193 nm Ultraviolet Photodissociation and Ultra High Resolution Tandem Mass Spectrometry. J Am Soc Mass Spectrom. 2017;28(7):1406-1419.
9. West H, Reid G E, Hybrid 213 nm photodissociation of cationized sterol lipid ions yield[m]+. Radical products for improved structural characterization using multistage tandem mass spectrometry. Anal Chim Acta, 2021, 1141:100-109.
10. Buré C, Le Sénéchal C, Macias L, Tokarski C, Vilain S, Brodbelt JS. Characterization of Isomers of Lipid A from Pseudomonas aeruginosa PAO1 by Liquid Chromatography with Tandem Mass Spectrometry with Higher-Energy Collisional Dissociation and Ultraviolet Photodissociation. Anal Chem. 2021;93(9):4255-4262.
11. Crittenden CM , Herrera CM , Williams PE , et al. Mapping phosphate modifications of substituted lipid A via a targeted MS3 CID/UVPD strategy. Analyst. 2018;143(13):3091-3099.
12. Macias L A, Feider C L, Eberlin L S, et al., Hybrid 193 nm ultraviolet photodissociation mass spectrometry localizes cardiolipin unsaturations. Anal Chem, 2019, 91:12509-12516.
13. Macias LA, Brodbelt JS. Enhanced Characterization of Cardiolipins via Hybrid 193 nm Ultraviolet Photodissociation Mass Spectrometry. Anal Chem. 2022;94(7):3268-3277.
14. Klein DR, Blevins MS, Macias LA, Douglass MV, Trent MS, Brodbelt JS. Localization of Double Bonds in Bacterial Glycerophospholipids Using 193 nm Ultraviolet Photodissociation in the Negative Mode. Anal Chem. 2020;92(8):5986-5993.
15. Klein D R, Feider C L, Garza K Y, et al., Desorption electrospray ionization coupled with ultraviolet photodissociation for characterization of phospholipid isomers in tissue sections. Anal Chem, 2018, 90:10100-10104.
欢迎致电400-099-8212联系我们
如需合作转载本文,请文未留言。

